遮罩陣列#
您將會做什麼#
使用 NumPy 的遮罩陣列模組來分析 COVID-19 數據並處理遺失值。
您將會學到什麼#
您將會理解什麼是遮罩陣列以及它們如何被建立
您將會看到如何存取和修改遮罩陣列的數據
您將能夠判斷在您的某些應用程式中何時適合使用遮罩陣列
您需要的東西#
Python 的基本熟悉度。如果您想複習一下記憶,請查看 Python 教學文件。
NumPy 的基本熟悉度
為了在您的電腦上執行繪圖,您需要 matplotlib。
什麼是遮罩陣列?#
考慮以下問題。您有一個包含遺失或無效條目的數據集。如果您要對此數據進行任何種類的處理,並且想要跳過或標記這些不需要的條目,而不是直接刪除它們,您可能必須使用條件語句或以某種方式過濾您的數據。numpy.ma 模組提供與 NumPy ndarrays 相同的部分功能,但增加了結構以確保無效條目不會用於計算。
出自 參考指南
遮罩陣列是標準 numpy.ndarray 和遮罩的組合。遮罩可以是
nomask,表示關聯陣列沒有無效值,或者是一個布林陣列,用於確定關聯陣列的每個元素的值是否有效。當遮罩的元素為False時,關聯陣列的對應元素是有效的,並稱為未遮罩。當遮罩的元素為True時,關聯陣列的對應元素被稱為已遮罩(無效)。
我們可以將 MaskedArray 視為以下項目的組合
數據,作為任何形狀或數據類型的常規
numpy.ndarray;與數據形狀相同的布林遮罩;
fill_value,一個可用於替換無效條目的值,以便返回標準numpy.ndarray。
它們在何時會很有用?#
在某些情況下,遮罩陣列可能比僅僅消除陣列的無效條目更有用
當您想要保留您遮罩的值以供稍後處理,而無需複製陣列時;
當您必須處理許多陣列,每個陣列都有自己的遮罩時。如果遮罩是陣列的一部分,您可以避免錯誤,並且程式碼可能更精簡;
當您對於遺失或無效值有不同的標記,並且希望保留這些標記而不替換原始數據集中的標記,但將它們從計算中排除時;
如果您無法避免或消除遺失值,但不想在您的操作中處理 NaN(非數字) 值。
遮罩陣列也是一個好主意,因為 numpy.ma 模組還附帶了大多數 NumPy 通用函數 (ufuncs) 的特定實作,這表示您仍然可以對遮罩數據應用快速向量化函數和操作。然後輸出會是一個遮罩陣列。我們將在下面看到一些關於這如何在實務中運作的範例。
使用遮罩陣列查看 COVID-19 數據#
從 Kaggle 可以下載一個數據集,其中包含關於 2020 年初 COVID-19 爆發的初始數據。我們將查看此數據的一小部分子集,包含在 who_covid_19_sit_rep_time_series.csv 檔案中。(請注意,此檔案已在 2020 年底的某個時間點被替換為沒有遺失數據的版本。)
import numpy as np
import os
# The os.getcwd() function returns the current folder; you can change
# the filepath variable to point to the folder where you saved the .csv file
filepath = os.getcwd()
filename = os.path.join(filepath, "who_covid_19_sit_rep_time_series.csv")
數據檔案包含不同類型的數據,並且組織如下
第一列是一個標頭行,(主要)描述了後續列中每列的數據,並且從第四列開始,標頭是觀察日期。
第二到第七列包含摘要數據,其類型與我們將要檢查的數據類型不同,因此我們需要將其從我們將要處理的數據中排除。
我們希望處理的數值數據從第 4 行、第 8 列開始,並從那裡延伸到最右邊的列和最下方的列。
讓我們探索此檔案中前 14 天記錄的數據。為了從 .csv 檔案收集數據,我們將使用 numpy.genfromtxt 函數,確保我們僅選擇包含實際數字的列,而不是包含位置數據的前四列。我們也跳過此檔案的前 6 列,因為它們包含我們不感興趣的其他數據。另外,我們將提取關於此數據的日期和位置資訊。
# Note we are using skip_header and usecols to read only portions of the
# data file into each variable.
# Read just the dates for columns 4-18 from the first row
dates = np.genfromtxt(
filename,
dtype=np.str_,
delimiter=",",
max_rows=1,
usecols=range(4, 18),
encoding="utf-8-sig",
)
# Read the names of the geographic locations from the first two
# columns, skipping the first six rows
locations = np.genfromtxt(
filename,
dtype=np.str_,
delimiter=",",
skip_header=6,
usecols=(0, 1),
encoding="utf-8-sig",
)
# Read the numeric data from just the first 14 days
nbcases = np.genfromtxt(
filename,
dtype=np.int_,
delimiter=",",
skip_header=6,
usecols=range(4, 18),
encoding="utf-8-sig",
)
在包含在 numpy.genfromtxt 函數呼叫中,我們為每個數據子集選擇了 numpy.dtype(整數 - numpy.int_ - 或字串 - numpy.str_)。我們也使用了 encoding 參數來選擇 utf-8-sig 作為檔案的編碼(在 官方 Python 文件 中閱讀更多關於編碼的資訊)。您可以從 參考文件 或從 Basic IO 教學文件 中閱讀更多關於 numpy.genfromtxt 函數的資訊。
探索數據#
首先,我們可以繪製我們擁有的整個數據集,看看它看起來像什麼。為了獲得可讀的圖表,我們僅選擇一些日期以顯示在我們的 x 軸刻度中。另請注意,在我們的繪圖命令中,我們使用 nbcases.T(nbcases 陣列的轉置),因為這表示我們將把檔案的每一列繪製為一條單獨的線。我們選擇繪製虛線(使用 '--' 線條樣式)。有關此的更多資訊,請參閱 matplotlib 文件。
import matplotlib.pyplot as plt
selected_dates = [0, 3, 11, 13]
plt.plot(dates, nbcases.T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020")
Text(0.5, 1.0, 'COVID-19 cumulative cases from Jan 21 to Feb 3 2020')
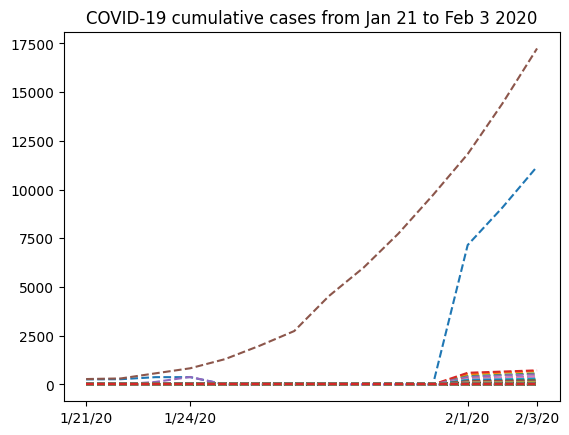
從 1 月 24 日到 2 月 1 日,圖表的形狀很奇怪。了解這些數據來自哪裡會很有趣。如果我們查看我們從 .csv 檔案中提取的 locations 陣列,我們可以看到我們有兩列,其中第一列將包含地區,第二列將包含國家名稱。但是,只有前幾列包含第一列的數據(中國的省份名稱)。在此之後,我們只有國家名稱。因此,將來自中國的所有數據分組到單行中是有道理的。為此,我們將僅從 nbcases 陣列中選擇 locations 陣列的第二個條目對應於中國的列。接下來,我們將使用 numpy.sum 函數來對所有選定的列求和 (axis=0)。另請注意,第 35 列對應於每個日期的整個國家的總計數。由於我們想要從省份數據中自行計算總和,因此我們必須先從 locations 和 nbcases 中刪除該列
totals_row = 35
locations = np.delete(locations, (totals_row), axis=0)
nbcases = np.delete(nbcases, (totals_row), axis=0)
china_total = nbcases[locations[:, 1] == "China"].sum(axis=0)
china_total
array([ 247, 288, 556, 817, -22, -22, -15, -10, -9,
-7, -4, 11820, 14410, 17237])
這個數據有些問題 - 我們不應該在累積數據集中有負值。發生什麼事了?
遺失數據#
查看數據,這是我們發現的:有一段時間有遺失數據
nbcases
array([[ 258, 270, 375, ..., 7153, 9074, 11177],
[ 14, 17, 26, ..., 520, 604, 683],
[ -1, 1, 1, ..., 422, 493, 566],
...,
[ -1, -1, -1, ..., -1, -1, -1],
[ -1, -1, -1, ..., -1, -1, -1],
[ -1, -1, -1, ..., -1, -1, -1]])
我們看到的所有 -1 值都來自 numpy.genfromtxt 嘗試從原始 .csv 檔案中讀取遺失數據。顯然,我們不希望將遺失數據計算為 -1 - 我們只是想跳過這個值,這樣它就不會干擾我們的分析。在導入 numpy.ma 模組後,我們將建立一個新的陣列,這次遮罩無效值
from numpy import ma
nbcases_ma = ma.masked_values(nbcases, -1)
如果我們查看 nbcases_ma 遮罩陣列,這就是我們擁有的
nbcases_ma
masked_array(
data=[[258, 270, 375, ..., 7153, 9074, 11177],
[14, 17, 26, ..., 520, 604, 683],
[--, 1, 1, ..., 422, 493, 566],
...,
[--, --, --, ..., --, --, --],
[--, --, --, ..., --, --, --],
[--, --, --, ..., --, --, --]],
mask=[[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False],
[ True, False, False, ..., False, False, False],
...,
[ True, True, True, ..., True, True, True],
[ True, True, True, ..., True, True, True],
[ True, True, True, ..., True, True, True]],
fill_value=-1)
我們可以看到這是一種不同類型的陣列。如簡介中所述,它具有三個屬性(data、mask 和 fill_value)。請記住,mask 屬性對於對應於無效數據的元素具有 True 值(在 data 屬性中以兩個破折號表示)。
讓我們嘗試看看數據的外觀,排除第一列(來自中國湖北省的數據),以便我們可以更仔細地查看遺失數據
plt.plot(dates, nbcases_ma[1:].T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020")
Text(0.5, 1.0, 'COVID-19 cumulative cases from Jan 21 to Feb 3 2020')
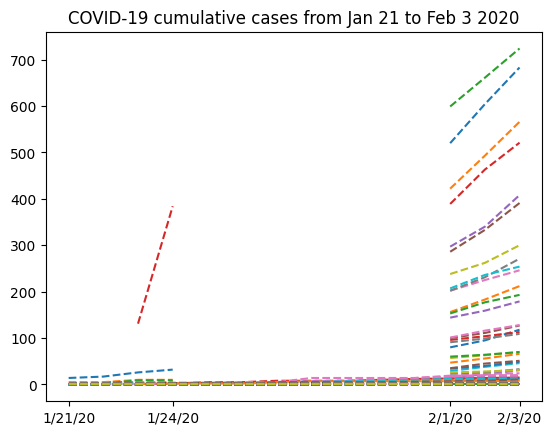
現在我們的數據已被遮罩,讓我們嘗試總結中國的所有病例
china_masked = nbcases_ma[locations[:, 1] == "China"].sum(axis=0)
china_masked
masked_array(data=[278, 309, 574, 835, 10, 10, 17, 22, 23, 25, 28, 11821,
14411, 17238],
mask=[False, False, False, False, False, False, False, False,
False, False, False, False, False, False],
fill_value=999999)
請注意,china_masked 是一個遮罩陣列,因此它具有與常規 NumPy 陣列不同的數據結構。現在,我們可以透過使用 .data 屬性直接存取其數據
china_total = china_masked.data
china_total
array([ 278, 309, 574, 835, 10, 10, 17, 22, 23,
25, 28, 11821, 14411, 17238])
這樣更好了:不再有負值。但是,我們仍然可以看到,在某些日子裡,累積病例數似乎下降了(例如,從 835 例降至 10 例),這與「累積數據」的定義不符。如果我們更仔細地查看數據,我們可以發現在中國大陸遺失數據的期間,香港、台灣、澳門和中國「未指明」地區有有效數據。也許我們可以從中國的病例總和中刪除這些,以更好地了解數據。
首先,我們將識別中國大陸地區的位置索引
china_mask = (
(locations[:, 1] == "China")
& (locations[:, 0] != "Hong Kong")
& (locations[:, 0] != "Taiwan")
& (locations[:, 0] != "Macau")
& (locations[:, 0] != "Unspecified*")
)
現在,china_mask 是一個布林值陣列(True 或 False);我們可以使用遮罩陣列的 ma.nonzero 方法檢查索引是否是我們想要的
china_mask.nonzero()
(array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19, 20, 21, 22, 23, 25, 26, 27, 28, 29, 31, 33]),)
現在我們可以正確地總結中國大陸的條目
china_total = nbcases_ma[china_mask].sum(axis=0)
china_total
masked_array(data=[278, 308, 440, 446, --, --, --, --, --, --, --, 11791,
14380, 17205],
mask=[False, False, False, False, True, True, True, True,
True, True, True, False, False, False],
fill_value=999999)
我們可以將數據替換為此資訊並繪製新圖表,重點關注中國大陸
plt.plot(dates, china_total.T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China")
Text(0.5, 1.0, 'COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China')
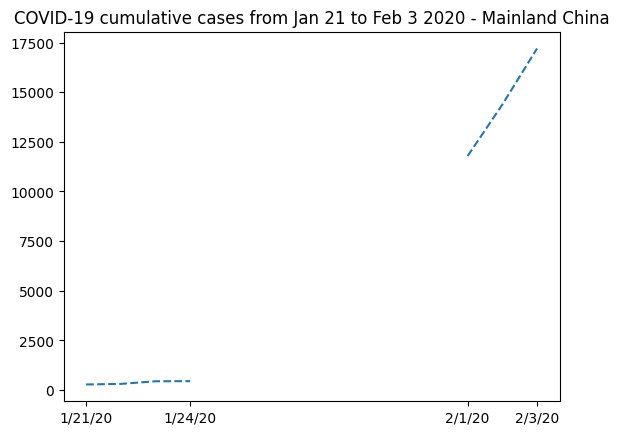
很明顯,遮罩陣列是這裡的正確解決方案。如果沒有錯誤地描述曲線的演變,我們就無法表示遺失數據。
擬合數據#
我們可以想到的一種可能性是內插遺失數據以估計 1 月下旬的病例數。觀察到我們可以使用 .mask 屬性選擇遮罩元素
china_total.mask
invalid = china_total[china_total.mask]
invalid
masked_array(data=[--, --, --, --, --, --, --],
mask=[ True, True, True, True, True, True, True],
fill_value=999999,
dtype=int64)
我們也可以使用此遮罩的邏輯否定來存取有效條目
valid = china_total[~china_total.mask]
valid
masked_array(data=[278, 308, 440, 446, 11791, 14380, 17205],
mask=[False, False, False, False, False, False, False],
fill_value=999999)
現在,如果我們想為此數據建立一個非常簡單的近似值,我們應該考慮無效條目周圍的有效條目。因此,首先讓我們選擇數據有效的日期。請注意,我們可以使用 china_total 遮罩陣列中的遮罩來索引日期陣列
dates[~china_total.mask]
array(['1/21/20', '1/22/20', '1/23/20', '1/24/20', '2/1/20', '2/2/20',
'2/3/20'], dtype='<U7')
最後,我們可以使用 numpy.polynomial 套件的擬合功能來建立一個最適合數據的三次多項式模型
t = np.arange(len(china_total))
model = np.polynomial.Polynomial.fit(t[~china_total.mask], valid, deg=3)
plt.plot(t, china_total)
plt.plot(t, model(t), "--")
[<matplotlib.lines.Line2D at 0x77d35cec04c0>]
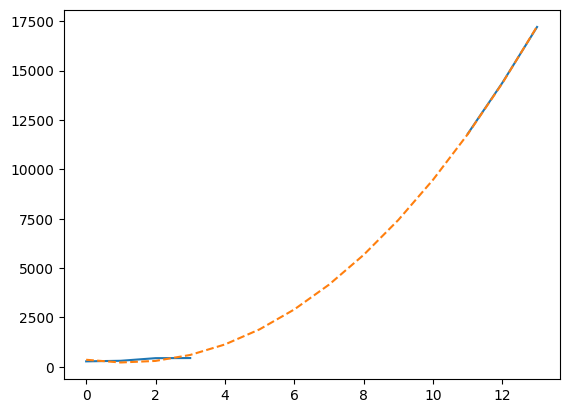
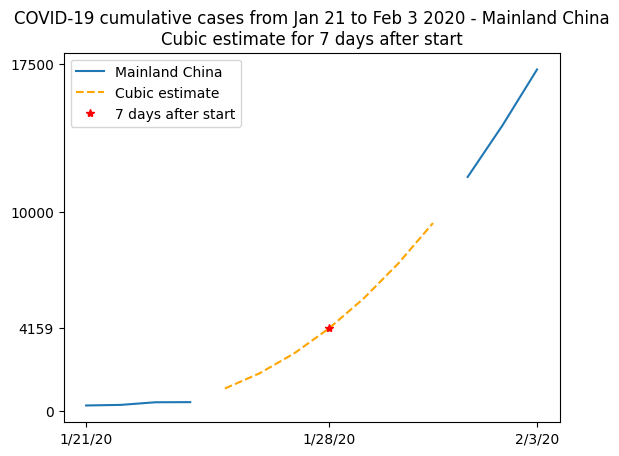
此圖表不是那麼容易閱讀,因為線條似乎彼此重疊,因此讓我們在更精細的圖表中進行總結。我們將在可用時繪製真實數據,並顯示不可用數據的三次擬合,使用此擬合來計算 2020 年 1 月 28 日(記錄開始後 7 天)觀察到的病例數估計值
plt.plot(t, china_total)
plt.plot(t[china_total.mask], model(t)[china_total.mask], "--", color="orange")
plt.plot(7, model(7), "r*")
plt.xticks([0, 7, 13], dates[[0, 7, 13]])
plt.yticks([0, model(7), 10000, 17500])
plt.legend(["Mainland China", "Cubic estimate", "7 days after start"])
plt.title(
"COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China\n"
"Cubic estimate for 7 days after start"
)
Text(0.5, 1.0, 'COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China\nCubic estimate for 7 days after start')
實務應用#
將
-1新增到遺失數據對於numpy.genfromtxt來說不是問題;在這種特殊情況下,將遺失值替換為0可能還可以,但稍後我們將看到這遠非通用的解決方案。此外,可以使用usemask參數呼叫numpy.genfromtxt函數。如果usemask=True,numpy.genfromtxt會自動傳回一個遮罩陣列。
延伸閱讀#
本教學文件中未涵蓋的主題可以在文件中找到
參考文獻#
Ensheng Dong, Hongru Du, Lauren Gardner, An interactive web-based dashboard to track COVID-19 in real time, The Lancet Infectious Diseases, Volume 20, Issue 5, 2020, Pages 533-534, ISSN 1473-3099, https://doi.org/10.1016/S1473-3099(20)30120-1.
